FastKing gDNA Dispelling RT SuperMix
Fitur
Cepat: Satu langkah untuk menyelesaikan penghapusan genom dan transkripsi balik yang efisien dalam waktu 18 menit dengan hanya menambahkan template.
Efisiensi tinggi: Transkriptase balik dimodifikasi dengan motif hidrofobik, dengan efisiensi RT melebihi 95%.
Sederhana dan mudah: DNase termosensitif eksklusif memiliki efek cepat, efisiensi tinggi dengan waktu reaksi yang lebih singkat, dan tidak akan memengaruhi cDNA.
Spesifikasi
Jenis: Transkriptase balik yang dimodifikasi gen, gDNase
Prosedur: Satu langkah (penghapusan DNA genom dan RT)
efisiensi RT: >95%
Templat: 1 ng- 2 g
Waktu operasi: ~18 menit
Aplikasi: cDNA yang ditranskripsikan terbalik dapat digunakan dalam PCR konvensional, PCR waktu nyata, konstruksi perpustakaan cDNA, SAGE (Analisis Serial Ekspresi Gen), ekstensi primer dan eksperimen konvensional lainnya.
Semua produk dapat disesuaikan untuk ODM/OEM. Untuk detailnya,silahkan klik Layanan yang Disesuaikan (ODM/OEM)
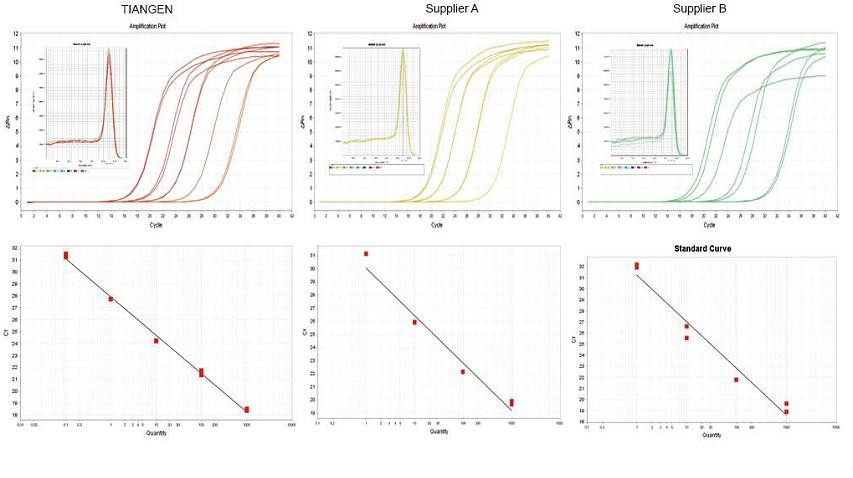 |
Contoh percobaan 1. cDNA disintesis menggunakan reagen kuantitatif terbalik satu langkah dari TIANGEN FastKing gDNA Dispelling RT SuperMix, produk yang relevan dari Pemasok A dan Pemasok B masing-masing. Deteksi gen RN5 mencit menggunakan TIANGEN Talent qPCR PreMix (SYBR Green), dan dianalisis kurva amplifikasi, kurva leleh, dan kurva standar. Hasilnya menunjukkan bahwa TIANGEN FastKing gDNA Dispelling RT SuperMix memiliki nilai Ct kuantitatif tertinggi setelah transkripsi balik dan ketahanan stres yang sangat baik, dan memiliki keunggulan yang jelas untuk template dengan residu pengotor tinggi. |
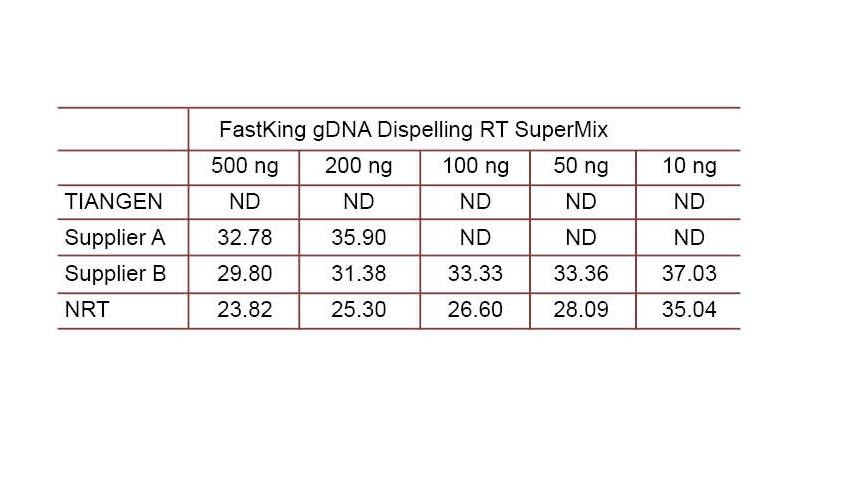 |
Contoh eksperimental 2. cDNA disintesis menggunakan reagen kuantitatif terbalik satu langkah dari TIANGEN FastKing gDNA Dispelling RT SuperMix, masing-masing produk yang relevan dari Pemasok A dan Pemasok B. Deteksi gen HsG manusia menggunakan TIANGEN Talent qPCR PreMix (SYBR Green), dan tambahkan secara manual berbagai konsentrasi DNA genom untuk mendeteksi kemampuan penghilangan gDNA dari berbagai reagen. Hasil Ct menunjukkan bahwa TIANGEN FastKing gDNA Dispelling RT SuperMix memiliki kemampuan yang sangat baik untuk menghilangkan DNA genom. Hingga 500 ng residu DNA genom dapat dihilangkan dengan sempurna tanpa mempengaruhi hasil. ND: Tidak terdeteksi. NRT: Deteksi campuran tanpa transkripsi terbalik. |
A-1 RNA terdegradasi
——Memurnikan RNA berkualitas tinggi tanpa kontaminasi. Bahan dari mana RNA diekstraksi harus sesegar mungkin untuk mencegah degradasi RNA. Analisis integritas RNA pada gel terdenaturasi sebelum reaksi RT. Setelah ekstraksi RNA, itu harus disimpan dalam formamida 100%. Jika RNase inhibitor digunakan, suhu pemanasan harus <45°C, dan pH harus kurang dari 8,0, jika tidak, inhibitor akan melepaskan semua RNase yang terikat. Selain itu, inhibitor RNase harus ditambahkan dalam larutan yang mengandung 0,8 mM DTT.
A-2 RNA mengandung penghambat reaksi transkripsi terbalik
——Inhibitor transkripsi terbalik termasuk SDS, EDTA, gliserol, natrium pirofosfat, spermidine, formamida, garam guanidin, dll. Campurkan RNA kontrol dengan sampel, dan bandingkan hasilnya dengan reaksi RNA kontrol untuk memeriksa apakah ada inhibitor. Cuci presipitasi RNA dengan 70% (v/v) etanol untuk menghilangkan inhibitor.
A-3 Tidak cukupnya anil primer yang digunakan untuk mensintesis untai pertama cDNA
——Tentukan bahwa suhu annealing cocok untuk primer yang digunakan dalam percobaan. Untuk heksamer acak, disarankan untuk menjaga suhu pada 25 ° C selama 10 menit sebelum mencapai suhu reaksi. Untuk primer spesifik gen (GSP), coba GSP lain, atau beralih ke oligo(dT) atau heksamer acak.
A-4 Sejumlah kecil RNA awal
——Meningkatkan jumlah RNA. Untuk sampel RNA kurang dari 50 ng, 0,1 g hingga 0,5 g asetil BSA dapat digunakan dalam sintesis cDNA untai pertama
A-5 Urutan target tidak diekspresikan dalam jaringan yang dianalisis.
——Coba tisu lain.
Reaksi PCR A-6 gagal
——Untuk RT-PCR dua langkah, cetakan cDNA pada langkah PCR tidak boleh melebihi 1/5 volume reaksi.
A-1 Anil primer dan template non-spesifik
—— Ujung 3' primer tidak boleh mengandung 2-3 dG atau dC. Gunakan primer spesifik gen dalam sintesis untai pertama alih-alih primer acak atau oligo(dT). Gunakan suhu anil yang lebih tinggi dalam beberapa siklus pertama, dan kemudian suhu anil yang lebih rendah. Gunakan hot-start Taq DNA polimerase untuk PCR untuk meningkatkan spesifisitas reaksi.
A-2 Desain primer gen spesifik yang buruk
——Ikuti prinsip yang sama untuk desain primer amplifikasi.
A-3 RNA terkontaminasi dengan DNA genom
——Perlakukan RNA dengan PCR-grade DNase I. Atur reaksi kontrol tanpa transkripsi balik untuk mendeteksi kontaminasi DNA.
A-4 Pembentukan dimer primer
——Desain primer tanpa urutan komplementer di ujung 3'.
A-5 Mg terlalu tinggi2+ konsentrasi
——Optimalkan Mg2+ konsentrasi untuk setiap kombinasi template dan primer
A-6 Terkontaminasi DNA asing
——Gunakan tip tahan aerosol dan enzim UDG.
A-1 Kandungan produk untai pertama terlalu tinggi
——Mengurangi jumlah produk untai pertama dalam langkah reaksi PCR konvensional.
A-2 Jumlah primer yang terlalu tinggi dalam reaksi PCR
——Kurangi input primer.
A-3 Terlalu banyak siklus
——Mengoptimalkan kondisi reaksi PCR dan mengurangi jumlah siklus PCR.
A-4 Suhu anil terlalu rendah
——Meningkatkan suhu anil untuk mencegah inisiasi dan ekstensi non-spesifik.
A-5 Amplifikasi non-spesifik fragmen oligonukleotida yang dihasilkan oleh degradasi DNase DNA ——Ekstrak RNA berkualitas tinggi untuk mencegah kontaminasi DNA.
RT-PCR adalah untuk membalikkan transkripsi RNA menjadi cDNA, dan kemudian menggunakan cDNA yang ditranskripsi terbalik sebagai template untuk reaksi PCR untuk memperkuat fragmen target. Pilih primer acak, Oligo dT dan primer spesifik gen sesuai dengan kondisi spesifik percobaan. Semua primer di atas dapat digunakan untuk mRNA sel eukariotik pendek tanpa struktur jepit rambut.
Primer acak: Cocok untuk RNA panjang dengan struktur jepit rambut, serta semua jenis RNA seperti rRNA, mRNA, tRNA, dll. Mereka terutama digunakan untuk reaksi RT-PCR dari template tunggal.
Oligo dT: Cocok untuk RNA dengan tailing PolyA (RNA prokariotik, eukariotik Oligo dT rRNA dan tRNA tidak memiliki ekor PolyA). Karena Oligo dT terikat pada ekor PolyA, kualitas sampel RNA harus tinggi, dan bahkan sejumlah kecil degradasi akan sangat mengurangi jumlah sintesis cDNA full-length.
Primer spesifik gen: Pelengkap untuk urutan template, cocok untuk situasi di mana urutan target diketahui.
Ada dua cara:
1. Metode referensi internal: Secara teori, cDNA adalah fragmen DNA dengan panjang yang berbeda, sehingga hasil elektroforesis adalah smear. Jika kelimpahan RNA rendah, tidak ada produk yang akan terlihat dalam elektroforesis, tetapi ini tidak berarti tidak ada produk yang akan diamplifikasi oleh PCR. Secara umum, referensi internal dapat digunakan untuk mendeteksi cDNA. Jika referensi internal memiliki hasil, kualitas cDNA pada dasarnya dapat dijamin (dalam beberapa kasus, jika fragmen gen target terlalu panjang, mungkin ada pengecualian).
2. Jika ada gen yang diketahui diamplifikasi oleh templat ini, itu dapat diverifikasi oleh primer gen ini. Amplifikasi referensi internal tidak berarti bahwa tidak ada masalah dengan cDNA. Karena referensi internal memiliki kelimpahan tinggi dalam cDNA, mudah untuk diperkuat. Jika cDNA terdegradasi sebagian karena berbagai alasan, dari sudut pandang probabilitas, hasil PCR dari gen target dengan kelimpahan rendah akan sangat terpengaruh. Sementara referensi internal masih berlimpah, amplifikasi mungkin tidak akan terpengaruh.
Sebagian mendegradasi RNA. Mendeteksi integritas dan memurnikan RNA
Isi RNA dari spesies yang berbeda mungkin berbeda, tetapi secara umum, RNA total yang diekstraksi harus mengandung dua pita 28S dan 18S yang jelas dalam elektroforesis gel, dan kecerahan pita sebelumnya harus dua kali lebih tinggi dari yang terakhir. Pita 5S menunjukkan bahwa RNA telah terdegradasi, dan kecerahannya sebanding dengan tingkat degradasi. Amplifikasi referensi internal yang berhasil tidak berarti tidak ada masalah dengan RNA, karena referensi internal berlimpah, RNA dapat diamplifikasi asalkan degradasinya tidak parah. OD260/OD280rasio RNA murni diukur dengan spektrofotometer harus antara 1,9 dan 2,1. Sejumlah kecil pengotor protein dalam RNA akan mengurangi rasio. Selama nilainya tidak terlalu rendah, RT tidak akan terpengaruh. Yang paling penting untuk RT adalah integritas RNA.
Perpanjangan gen referensi internal hanya dapat menunjukkan bahwa RT telah berhasil, tetapi belum tentu terkait dengan kualitas untai cDNA. Karena fragmen referensi internal umumnya berukuran kecil dan berekspresi tinggi, mereka lebih mudah berhasil dalam transkripsi balik. Namun, ukuran dan ekspresi gen target bervariasi dari gen ke gen. Kualitas cDNA tidak dapat dinilai hanya dengan referensi internal terutama untuk fragmen target yang lebih panjang dari 2 kb.
Beberapa sampel memiliki struktur sekunder yang kompleks, atau memiliki kandungan GC yang kaya, atau sangat berharga dengan kelimpahan yang rendah. Dalam kasus ini, transkriptase balik yang sesuai harus dipilih sesuai dengan ukuran fragmen target dan sampel. Untuk template RNA dengan konten GC tinggi dan struktur sekunder kompleks, sulit untuk membuka struktur sekunder pada suhu rendah, atau dengan reverse transcriptase umum. Untuk templat ini, Quant Reverse Transcriptase dapat dipilih, karena kinerja transkripsi baliknya jelas lebih baik daripada transkriptase terbalik seri M-MLV, yang dapat mentranskripsi balik berbagai templat RNA secara efisien dan mentranskripsikan RNA ke untai pertama cDNA secara maksimal. Saat menggunakan kit reverse transcriptase umum, sistem 20 l hanya dapat secara efektif membalikkan transkripsi 1 g total RNA. Harap perhatikan kapasitas RT maksimum kit. Jika template ditambahkan secara berlebihan, transkripsi balik akan mendukung RNA dengan kelimpahan tinggi. Oleh karena itu, lebih baik tidak melebihi kapasitas maksimum sistem.
A-1 Tentukan apakah RNA terdegradasi parah dan jika RT berhasil
Secara umum, alasan kegagalan amplifikasi referensi internal sering disebabkan oleh degradasi RNA yang serius. Alasan lain yang mungkin adalah kegagalan transkripsi terbalik. Referensi internal tidak dapat digunakan sebagai standar untuk menilai kualitas untai tunggal cDNA, tetapi dapat digunakan sebagai standar untuk menilai apakah transkripsi balik berhasil jika tidak ada masalah kualitas RNA. Hal terpenting dalam proses transkripsi balik adalah menjaga suhu konstan dan sistem reaksi konstan untuk meningkatkan efisiensi reaksi.
A-2 Tentukan apakah primer untuk memperkuat gen referensi internal dapat diandalkan dan jika ada masalah dengan reagen yang digunakan dalam PCR.
Untuk kuantifikasi relatif, RNA harus dikuantifikasi sebelum transkripsi terbalik, yang juga diperlukan dalam banyak kit transkripsi terbalik, misalnya, mengukur input RNA sebagai 1 g. Karena cDNA yang ditranskripsi balik adalah larutan campuran, termasuk RNA, oligo dT, enzim, dNTP, dan bahkan sedikit residu DNA, penyimpangan akan terjadi, sehingga tidak mungkin untuk mengukur cDNA secara akurat. Oleh karena itu, kuantifikasi RNA diperlukan. Mengingat efisiensi transkripsi terbalik adalah sama di antara sampel yang berbeda, jumlah cDNA yang diperoleh harus sama, dan analisis kuantitatif dapat menunjukkan perbandingan tingkat ekspresi gen yang berbeda dalam jumlah total RNA yang sama. Saat melakukan PCR kuantitatif fluoresensi relatif, cDNA kuantitatif mungkin tidak diperlukan setelah transkripsi balik karena gen referensi internal dapat bertindak sebagai referensi.
Ini terutama terkait dengan gen, dan transkripsi terbalik dari fragmen panjang tidak layak untuk sebagian besar gen. Pertama, efisiensi transkripsi balik jauh lebih rendah daripada PCR. Kedua, wilayah kaya GC dan struktur sekunder banyak gen membatasi transkripsi balik dan PCR. Akhirnya, fidelitas dan efisiensi amplifikasi PCR sulit dijamin pada saat yang bersamaan. Dalam proses transkripsi balik, tidak ada yang bisa menjamin untuk mendapatkan fragmen panjang untuk gen salinan rendah, terutama menggunakan oligo dT. Sedangkan untuk UTR 5' dengan GC lebih banyak, lebih sulit lagi. Oleh karena itu, masih merupakan metode yang masuk akal untuk membalikkan transkrip dengan primer acak, menemukan situs pembelahan alami dalam fragmen target, memperkuat segmen, dan kemudian melakukan pencernaan restriksi dan ligasi. Secara umum, sulit untuk secara langsung mengamplifikasi fragmen yang lebih besar dari 2 kb, tetapi tidak selalu mustahil untuk mendapatkan: 1.Pertama-tama, menjamin integritas RNA/mRNA, dan ekstraksi TRIZOL lebih disukai. 2.M-MLV RT-PCR kit dapat langsung digunakan. Memperpanjang waktu anil dan meningkatkan jumlah siklus dalam proses amplifikasi dengan benar. Sebagai alternatif, PCR bersarang dapat diterapkan, atau melakukan satu atau dua reaksi terlebih dahulu dengan denaturasi dan waktu ekstensi yang diperpanjang secara tepat sebelum amplifikasi PCR normal, yang dapat membantu memperpanjang fragmen. Perhatikan kesetiaan polimerase. 3.Long Taq dapat digunakan dalam PCR untuk mendapatkan hasil yang ideal. 4.Untuk aplikasi ekspresi protein, polimerase kesetiaan tinggi harus diterapkan.
Ada dua jenis reverse transcriptase yang ditawarkan oleh TIANGEN: Quant/King RTase dan TIANScript M-MLV. Perbedaan utama di antara mereka adalah jumlah input template. Quant adalah reverse transcriptase unik, yang berbeda dari M-MLV yang umum digunakan yang berasal dari virus leukemia murine Moloney. Quant adalah transkriptase terbalik efisiensi tinggi baru yang diekspresikan secara rekombinan oleh rekayasa Escherichia coli. Quant cocok untuk memperkuat 50 ng-2 g RNA dengan aktivitas transkripsi balik tinggi dan hasil tinggi. Dibandingkan dengan MMLV atau AMV biasa, karakteristik terbesar Quant adalah memiliki afinitas yang sangat kuat dengan template RNA dan dapat membalikkan template kompleks transkrip tanpa denaturasi suhu tinggi. Untuk template dengan konten GC yang lebih tinggi, efisiensi sebaliknya lebih tinggi. Namun, transkriptase balik ini memiliki aktivitas RNase H, yang dapat mempengaruhi panjang produk cDNA (cocok untuk templat <4,5 kb). Untuk transkripsi terbalik konvensional, TIANScript MMLV reverse transcriptase direkomendasikan. RTase ini adalah enzim yang dimodifikasi dengan aktivitas RNase H yang sangat lemah, yang cocok untuk sintesis cDNA yang panjang (> 5 kb).
Transkripsi balik satu langkah dan amplifikasi PCR diselesaikan dalam tabung yang sama tanpa membuka penutup tabung antara sintesis cDNA dan amplifikasi, yang berguna untuk mengurangi kontaminasi. Karena semua sampel cDNA yang diperoleh digunakan untuk amplifikasi, sensitivitasnya lebih tinggi, dengan minimal 0,01 pg dari total RNA. Untuk RTPCR satu langkah yang berhasil, primer spesifik gen umumnya digunakan untuk memulai sintesis cDNA. Metode dua langkah yaitu transkripsi balik dan amplifikasi PCR dilakukan dalam dua langkah. Transkripsi balik pertama dilakukan dari template RNA untuk mendapatkan cDNA, dan cDNA yang diperoleh dikenai satu atau lebih reaksi PCR yang berbeda. Metode dua langkah dapat menggunakan oligo(dT) atau primer acak untuk memandu sintesis untai pertama cDNA, dan dapat membalikkan transkripsi semua informasi mRNA dari sampel tertentu.
Kategori produk
MENGAPA MEMILIH KAMI
Sejak didirikan, pabrik kami telah mengembangkan produk kelas dunia pertama dengan mengikuti prinsip
kualitas pertama. Produk kami telah mendapatkan reputasi yang sangat baik di industri dan kepercayaan yang berharga di antara pelanggan baru dan lama..